La Inteligencia Artificial (IA) está de moda. Incluso se debate si un algoritmo nos quitará el puesto de trabajo o si es “ético”. La IA se aplica a cualquier aspecto de la sociedad moderna, desde la fabricación de automóviles hasta la publicidad en la web. Esto es debido a dos razones principales: la gran cantidad de datos que se pueden obtener gracias a internet y al desarrollo de nuevos algoritmos basados en el denominado “aprendizaje profundo” o Deep Learning. Esta transformación también ha afectado a la agricultura. Gracias a la disponibilidad de sensores, drones y tecnologías genómicas, hoy en día podemos hablar de “agricultura inteligente” o “de precisión”.
El objetivo de este proyecto es desarrollar algoritmos de aprendizaje profundo en algunos aspectos cruciales de la agricultura, como son la predicción genómica y la simulación por ordenador. El concepto de “predicción” es muy importante en numerosas actividades científicas e industriales. Por ejemplo ¿cuál es la probabilidad de que un cliente cambie de compañía telefónica si vive en tal distrito postal y tiene esta franja de edad? ¿cuál es la vida esperada de un electrodoméstico? O en agricultura, ¿cuál es la mejor variedad genética para este clima y en este suelo? ¿cuál será la producción lechera de esta vaca? Mejorar la capacidad de predicción en agricultura es clave para lograr una producción más sostenible y eficiente, y para adaptar las variedades al cambio climático.
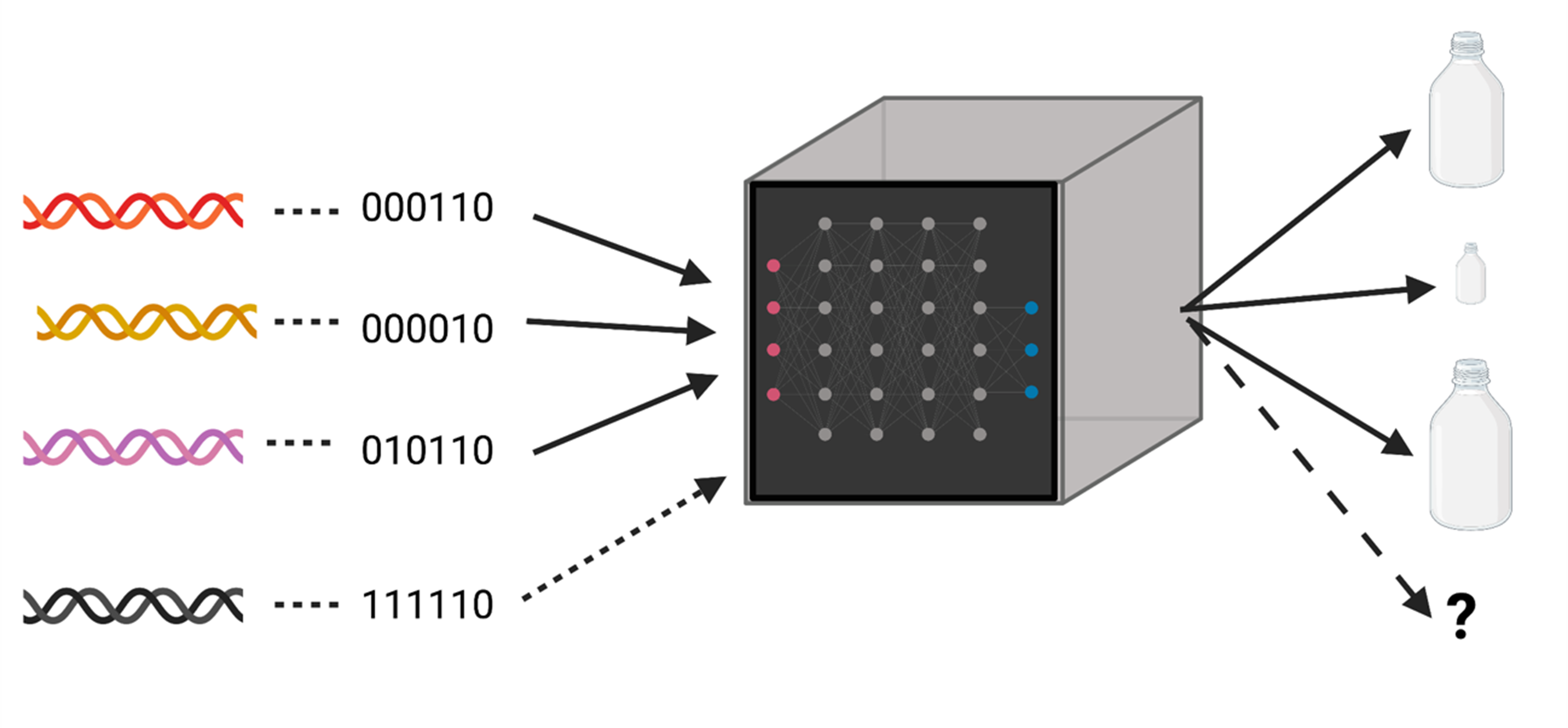
Gracias a su flexibilidad, el aprendizaje profundo se puede utilizar para resolver todos estos y muchos más problemas. Los detalles matemáticos son complicados, pero la idea es relativamente sencilla (Figura 1). En primer lugar, los ordenadores entienden solo números, una fotografía es un conjunto de números, en dos dimensiones si es blanco y negro o en tres si es en color. El sonido, o cualquier otro concepto, también son números. En segundo lugar, el algoritmo “aprende” con datos conocidos y validados. Por ejemplo, si queremos estudiar la mejor variedad genética en un ambiente, debemos conseguir primero ensayos de campo, en una diversidad de climas, con distintas variedades genéticas similares a las que queremos estudiar. La palabra “similar” es muy importante, quiere decir que el algoritmo solo “aprende” de lo que ve, no tiene “imaginación”. En tercer lugar, el algoritmo, si todo va bien, es capaz de descubrir patrones de variación no obvios que relacionan el clima y la variación genética con el rendimiento de la cosecha. Esto lo hace mediante transformaciones no lineales, complejas que se llaman “neuronas” en la jerga del aprendizaje profundo.
El segundo aspecto en el que incide nuestro proyecto es en el de “simulación” por ordenador. Tal como existen simuladores de vuelo donde los pilotos se entrenan, también la simulación es muy importante en agricultura, con el objetivo de recrear cuál sería la producción agrícola bajo determinadas condiciones. La simulación es a menudo el único enfoque para resolver numerosos problemas en genética y agricultura. En nuestro caso, queremos estudiar el efecto del microbioma en la salud y productividad de los animales. El microbioma es el conjunto de microorganismos que viven en el cuerpo de animales, por ejemplo, en el intestino. Hay evidencia abrumadora de que la flora intestinal se ve afectada y afecta a gran número de caracteres, como la absorción intestinal, diabetes, etc.
Aquí de nuevo las técnicas de aprendizaje profundo pueden ser muy útiles. En particular, estamos estudiando los modelos llamados “generativos”, tales como las “redes adversarias generativas” (GAN es su acrónimo inglés). El algoritmo subyacente es fascinante, y consiste en poner a competir dos redes neuronales, una que genera (simula) datos y otra que decide si el dato simulado es real o no. Ambas redes compiten entre sí para mejorar y al final se llega a un equilibrio llamado “minimax” donde el discriminador solo acierta la mitad de las veces, que es lo máximo posible cuando el dato simulado es indistinguible del real. Las apps del móvil que te hacen envejecer o te cambian de sexo son algunas de las aplicaciones de esta técnica. En nuestro caso, como ejemplo preliminar, hemos usado las GANs para simular frutos a partir de fotos de cantaloupe, melón piel de sapo y de otras hortalizas como el pepino (Figura 2). Como podemos observar, algunas imágenes son de gran realismo y reproducen la textura y la forma de frutas reales, pero en nuevas combinaciones.

Los resultados de este proyecto mejoraran las herramientas existentes de predicción genómica y simulación, lo que tiene una aplicación inmediata en la agricultura. Unas mejores herramientas predictivas, a su vez, aumentarán la eficiencia y precisión de la agricultura y contribuirán a paliar el impacto medioambiental. Sin embargo, el impacto de este proyecto se extiende más allá de estas aplicaciones específicas. Los "analistas de datos" y especialistas en inteligencia artificial se encuentran entre los profesionales más buscados, y contribuiremos a capacitar a jóvenes investigadores en esta área.

Miguel Pérez Enciso es Profesor de Investigación ICREA en el Centro de Investigación en Genómica Agrícola (CRAG) en el campus de la Universidad Autónoma de Barcelona. Es doctor en Genética por la Universidad Complutense de Madrid (1990). Trabajó en el Institut de Recerca i Tecnologia Agroalimentaria (IRTA) desde 1993 hasta 1999 y en el Institut National de la Recherche Agronomique (INRA) en Toulouse (Francia) desde 1999 hasta 2003. También es profesor a tiempo parcial en la Universidad Autónoma de Barcelona. Sus intereses actuales incluyen la aplicación de tecnologías de aprendizaje automático a la genómica en ganadería, humanos y las plantas, el uso de datos de secuencia para la selección genómica y el desarrollo de software. Estudia cómo los algoritmos pueden generar distribuciones muy complejas, como formas, comportamientos o abundancias microbianas.



